16S rRNA 유전자 분석을 이용한 지표수-지하수 혼합구간에 서식하는 배양성 세균의 다양성
초록
본 연구에서는 강원도 양구군 해안면에서 채취한 지표수, 지하수-지표수 혼합구간(hyporheic zones) 및 지하수 시료에 서식하는 배양성 미생물을 확인하기 위해 16S rRNA 유전자 PCR 방법을 사용하여 동정하는 실험을 진행하였다. 배양성 미생물의 세균수를 측정한 결과, 지표수-지하수 혼합구간에서 2.5×105 CFU/ml로 지표수와 지하수에 비해 10배 이상의 세균수가 확인되었다. 또한 호기성, 혐기성 미생물을 분리하여 세균수를 측정한 결과에서도 혐기성 세균보다 호기성 세균이 10-100배 더 많이 확인 되었다. 16S rRNA 유전자 염기서열 분석을 통한 동정 결과 혼합구간 호기성 세균 40개 속, 혐기성 세균 21개 속으로 동정 되었으며, 호기성 세균은 속수준에서 Pseudomonas 속이 약 29.3%로 우점하였고, Flavobacterium 속이 약 15.3%로 차우점 하였다. 지표수, 지하수에서 분리된 속을 제외하고, 혼합구간에서만 나타난 속은 총 호기성 31개, 혐기성 16개 속으로 확인되었다. 지표수-지하수 혼합구간 시료에서 Pyrosequencing 방법으로 획득한 메타지노믹 자료와 본 연구를 통해 획득한 배양성 미생물과 비교분석 하면 혼합구간에 서식하는 미생물들의 생리적인 특성 및 기능과 역할에 대해 보다 명확한 기초 자료를 제공할 수 있을 것이며 보다 정량적인 기초자료를 제공할 수 있을 것으로 여겨진다.
Abstract
This study identified 16S rRNA genes using PCR to find out culturable microorganisms living in the samples of surface water, surface water-groundwater exchange zones (hyporheic zones), ground water collected in the Haean bain, Yanggu-gun, Gangwon-do. Bacterial count of culturable microorganisms was 2.5×105 CFU/ml in the hyporheic water, which was over 10 times higher than those in the surface water and the ground water. Moreover, measurements of bacterial counts by separation into aerobic and anaerobic microorganisms found 10-100 times more aerobic bacteria than anaerobic bacteria. Identification through sequencing of 16S rRNA gene resulted in 40 genera of aerobic bacteria and 21 genera of anaerobic bacteria in the hyporheic water. Of the aerobic bacteria, the Pseudomonas genus was most dominant with about 29.3% in proportion, followed by the Flavobacterium genus with about 15.3%. Excluding genera separated from the surface water and the ground water, a total of 31 aerobic genera and 16 anaerobic genera were identified in the hyporheic water. It is expected that a comparative analysis between metagenomics data obtained from samples in the hyporheic water by pyrosequencing and culturable microorganisms of the present study should be able to provide more solid fundamental data on physiological characteristics and roles of microorganisms living in hyporheic zones and would enable to conduct more quantitative study.
Keywords:
surface water-groundwater exchange zones (hyporheic), culturable bacteria, 16S rRNA키워드:
지표수-지하수 혼합구간 (하이포릭), 배양성 세균, 16S rRNA1. 서 론
수계 환경에서 미생물은 세균, 고세균, 조류, 진균 및 플랑크톤 등을 포함하며, 하상(streambed)에 정착하거나 바이오필름을 생성하는 것으로 보고되었다(Mugani et al., 2015). 지하수와 지표수 사이에 존재하는 혼합구간(Hyporheic zone)은 생지화학적으로 다양한 수환경 생태계를 가지며, 탄소, 영양분 및 다양한 물질에 영향을 받는 것으로 알려져 있다(Graham et al., 2016). 지표수-지하수 혼합구간에 대한 연구는 1) 미생물들의 호흡이 가장 많이 발생하는 구간(Grimm and Fisher, 1984), 2) 혼합구간에서의 질화 및 탈질화 과정에 따라 전체 무기태 질소의 양의 변화가 발생(Triska et al., 1993), 3) 지표수가 높은 미생물 밀도를 가질 때 하강류가 발생하게 되고 반면 지하수는 상승하게 되어 지표수-지하수 혼합구간 형성(Hendricks, 1993) 등의 다양한 관점에서 이뤄지고 있다(Storey et al., 1999).
일반적으로 환경시료에 대한 미생물 연구방법으로 유전체의 염기서열을 고속으로 분석하는 차세대염기서열분석을 많이 사용한다. 이 분석방법에는 실시간 염기서열분석(Single-molecule real-time sequencing) (Murray et al., 2012), 이온 토런트 염기서열분석(Ion Torrent sequencing) (Quail et al., 2012), 파이로시퀀싱(pyrosequencing) (Kim et al., 2018a), 염기서열 합성 분석(Sequencing by synthesis) (van Vliet, 2010), 염기서열 묶음 분석(Sequencing by ligation) (Huang et al., 2012) 등의 비배양성 미생물 분석 방법이 주로 이뤄지고 있으며 전체 세균의 다양성(Feris et al., 2003a) 및 특정 기능성 미생물의 분석(Pei et al., 2010)에 사용되고 있다. 또한 변성 구배 젤을 이용한 전기영동으로 DNA 염기서열의 구조적 차이를 구별하는 변성 구배 젤 전기영동(Denaturing Gradient Gel Electrophoresis) (Iwamoto et al., 2000) 방법도 사용한다. 반면 배양성 미생물의 분석은 지표수, 해수, 지하수 시료에 대한 연구가 주로 이뤄지고 있으며, 지표수-지하수 혼합구간에서의 미생물 연구는 적은 것으로 보고되었다(Hendricks, 1993).
분자생물학적 분석기법의 발달로 여러 환경에 존재하는 다양한 미생물의 종들을 구별하고 동정(Identification)할 수 있게 되었는데, 대부분 ribosomal RNA 유전자서열의 차이를 이용하여 미생물의 군집구조 및 미생물의 종류를 파악할 수 있었다(Lee et al., 2018; Kim and Lee, 2019). 특히 지표수-지하수 혼합구간은 지표수 및 지하수에 서식하는 미생물에 비하여 생리적 및 물질대사적 다양성을 반영하는데 매우 이질적인 환경을 가지고 있다(Kim and Lee, 2019). 앞서 언급한 파이로시퀀싱 방법을 이용한 혼합구간 내 미생물의 다양성 결과는 지표수 및 지하수 결과에 비해 높게 나타난다(Kim et al., 2015; Kim et al., 2018a). 이와 같이 미생물의 다양성은 유전적 다양성으로 대변된다. 하지만 차세대염기서열 분석 방법 중 하나인 파이로시퀀싱 방법은 활성이 없는 죽은 미생물까지도 포함한다는 단점이 있고, 장비 및 분석비가 고가이다. 또한 다양한 미생물의 존재를 확인하고 그들의 역할을 단순히 예측하는 정도에서 그치므로 각각의 미생물들의 생리적인 특성 및 기능과 역할에 대해 연구하기에는 미흡한 점이 많다(Bae, 2011). 그러므로 보다 정량적인 연구를 위해서는 지표수-지하수 혼합구간의 시료에서 배양성 미생물에 대한 추가적인 연구가 필요하다.
혼합구간은 지표수와 지하수가 점진적으로 변화하는 점이구간으로 하천 하상 아래에 존재하며, 지하 수 cm에서 수 km까지 퍼져있는 구간이다(Feris et al., 2003b; Kim et al., 2014). 지하수 와 지표수 사이에서 완충작용을 하며, 영양분의 유입 또는 저장을 하고, 지표수에서 유입된 유기물의 저감이 일어나는 중요한 장소로 알려져 있다(Jones et al., 1995). 혼합구간은 높은 생물학적 활동이 일어나는 곳으로 알려져 있으며, 하천 생태계의 영양분 공급지역으로 알려져 있다(Storey et al., 1999). 혼합구간 환경에 존재하는 미생물은 지표수 및 지하수에서 유발된 오염물질의 분해가 가능하다. 혼합구간의 토착미생물의 오염물질 저감능은 이곳에 서식하는 조류, 무척추동물, 중형저서동물 및 강기슭 식물 등 수 생태계를 구성하는 동식물에게 긍정적인 영향을 미치는 것으로 보고되었다(Barlocher and Murdoch, 1989; Pusch and Schwoerbel, 1994; Kim et al., 2018a). 혼합구간의 미생물들은 95% 이상이 지표수 생태계의 환경에 영향을 받으며, 생태학적으로 잘 알려져 있지 않다(Graham et al., 2016). 몇몇의 연구자가 혼합구간의 미생물에 대해 연구하였으나(Jeon et al., 2018; Kim et al., 2018a; Kim and Lee, 2019), 극히 일부만 연구되었고, 하천수, 호수 토양 및 해양생태계에 대한 연구가 주로 이뤄졌었다(Brunke and Gonser, 1997; Storey et al., 1999). 본 연구에서는 파이로시퀀싱 방법의 단점을 보완하고자 지표수, 지표수-지하수 혼합구간 그리고 지하수에서 서식하는 배양성 미생물을 확인하였다. 이를 확인하기 위해 16S rRNA 유전자 PCR을 통해 동정하는 실험을 진행하였다.
2. 연구 방법
2.1 연구지역
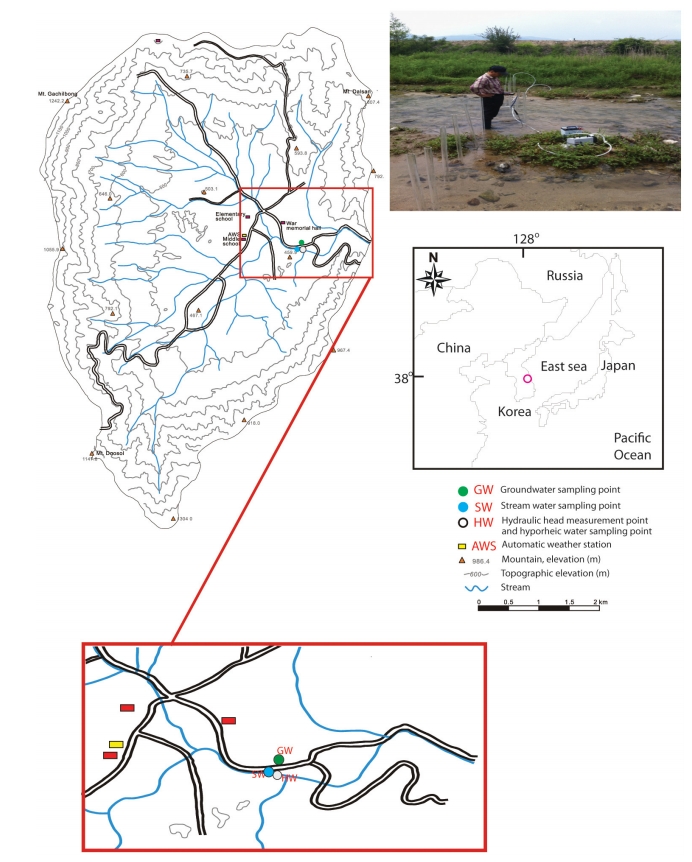
연구지역은 행정구역상 강원도 양구군 해안면으로 한반도의 정중앙에 위치한다. 해안면 일대는 가칠봉(1,242 m), 도솔산(1,147 m), 대우산(1,056 m) 및 달산령(807 m)과 같이 해발고도가 높은 험준한 산들로 외부가 둘러싸여 있고 내부는 평탄한 분지지형으로 해안분지라고 불린다(Lee, 2009; Kim et al., 2018b). 또는 펀치볼 마을이라고 하는데 6.25전쟁 당시 외국의 종군기자가 가칠봉에서 노을 진 분지가 칵테일 유리잔 속의 술 빛과 같고 화채그릇(Punch Bowl)과 닮았다 하여 이와 같은 지명이 붙여졌다.
해안 분지의 지질은 선캄브리아기 경기편마암 복합체로 규암, 편암 및 편마암으로 구성되며 이를 쥬라기 대보화강암이 관입하였다(Lee, 2009; Kim et al., 2018b). 쥬라기 대보화강암은 분지의 중앙에 광범위하게 분포하고 그 외륜은 선캄브리아기 변성퇴적암이 둘러싸고 있다(Won et al., 1987). 해안 분지의 성인은 차별침식에 의해 형성 되었다(Lee, 2009). 분지 내에는 성황천, 도솔천, 만대천과 함께 다수의 지천이 발달하였지만 분지 바깥쪽으로 유출하는 하천은 당물골 하나뿐이다. 그리하여 타 지역에 비하여 매우 단순한 수리구조를 가지고 있다. 하천뿐만 아니라 지하수 또한 이 하천으로 수렴하여 빠져 나간다(그림 1). 연구지역의 이러한 수리구조의 특성은 지하수와 하천간의 상호관계, 지표수-지하수 혼합구간에 서식하는 토착미생물 또는 혼합구간에서 오염물질의 유동 및 저감에 대한 연구에 최적이다(Lee, 2009; Kim et al., 2018a; Lee et al., 2019).
2.2 지표수-지하수 혼합구간의 시료채취 및 배양성 세균 분리
지표수, 지표수-지하수 혼합구간 그리고 지하수의 배양성 미생물 특성을 조사하기 위하여, 지표수 시료 1개(SW), 혼합구간(HW) 물시료 10개(P1~P10) 및 지하수 시료 1개(GW)를 채취하였다. 2016년 8월 5일(12개)과 2016년 12월 13일(12개) 총 24개 시료를 채취하였다. 혼합구간의 시료 채취를 위해 하천의 횡단면에 소형관정 10개를 설치하였다(그림 1). 혼합구간의 시료채취 기준은 하상의 깊이별 온도자료로 열전달 해석을 진행하여 혼합구간의 깊이를 추정한 Kim et al. (2014)이 사용한 방법을 사용하였다. 온도자료를 이용하여 계산된 깊이(9.2~14.9 cm)를 혼합구간이라고 판단하고 시료를 채취하였다. 시료 채취방법은 Kim et al. (2018a)에 기술된 내용을 따라 진행하였다. 시료는 아이스박스에 4℃를 유지하며 실험실로 옮긴 뒤, 배양성 미생물 특성 조사에 사용하였다. 채취한 시료는 단계희석법을 사용하여 희석 후 Reasoner's 2A agar (R2A agar, Oxoid, UK)배지에 도말하여 배양성 미생물을 분리하였다. 한편 혐기성 미생물을 분리하기 위하여 BD BBLTM CO2 generators (BD, USA)와 CO2 indicator (BD)를 사용하였다. 배양 조건은 25℃에서 2일간 배양 후 배양성 미생물의 집락 개수를 통해 세균수 (CFU/ml)를 측정하였다. 각 시료의 조건 당 최소 10개의 집락을 분리하였다.
2.3 호기성 및 혐기성 배양성 세균 동정
분리된 미생물의 동정을 위해 InstaGeneTM Matrix (Bio-rad, USA)를 이용하여 핵산을 추출하고, 16S rRNA 유전자 PCR을 진행하였다. PCR 반응에 사용한 프라이머는 universial primer인 27F, 1492R을 이용하였다. PCR 조성은 AccuPower® HotStart PCR PreMix (Bioneer, Korea), 주형 핵산 1 µl, 프라이머 2 µl (정방향 1 µl, 역방향 1 µl; 20 pmole/µl), nucleic acid free water 17 µl로 총 20 µl로 구성하였다. PCR 조건은 94℃ 1분 이후 94℃ 30초, 55℃ 30초, 72℃ 1분을 30번 반복 후 마지막으로 72℃ 5분간 반응시켰다. 1.2% agarose gel 상에 전기영동 하여 1.5 kb의 밴드를 확인하였다. 16S rRNA 유전자 증폭이 확인된 증폭 산물은 Macrogen (Korea)에 sequencing을 의뢰하여 염기서열 분석을 진행하였다. Sequencing 이후 분석된 염기서열은 Eztaxon program (Yoon et al., 2017)을 이용하여 분리된 세균의 미생물 동정을 진행하였다.
3. 결과 및 고찰
3.1 지표수-지하수 혼합구간의 배양성 세균의 특성
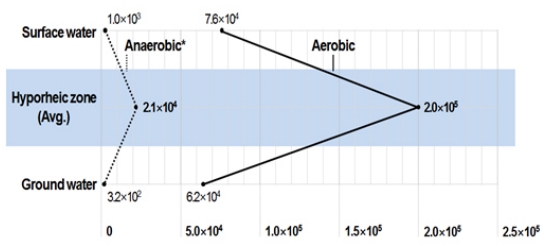
지표수, 혼합구간, 지하수에서 채취된 시료 24개의 세균수를 측정한 결과, 지표수와 지하수 보다 혼합구간에서 2.5×105 CFU/ml로 가장 많은 세균수가 확인되었다(표 1). 호기성 및 혐기성 세균수를 측정한 결과 대체적으로 호기성 세균수가 혐기성 세균수보다 최대 100배 정도 더 많은 것으로 확인되었다(표 1). 혼합구간 별 호기 및 혐기성 세균수를 측정한 결과, 호기성 세균은 2.2×104 – 5.6×105 CFU/ml, 혐기성 세균은 1.3×103 – 6.5×104 CFU/ml로 확인 되었다. 지표수에 비해 혼합구간의 호기성 미생물은 약2.7배, 혐기성 미생물은 약 19.9배 높게 나타났으며, 지하수에 비해 혼합구간의 호기성 미생물은 약 3.3배, 혐기성은 약 64.7배 높게 나타났다(그림 2). 1개 시료(P8)를 제외하고 전체적인 세균수는 지하수 및 지표수에 비해 높게 나타났으며, 호기(5.6×105 CFU/ml) 및 혐기성(6.5×104 CFU/ml) 모두 P6 시료에서 가장 높은 세균수가 확인되었다. 혐기성 미생물의 경우 상대적으로 P1, P3, P5 및 P6 시료에서 높은 세균수가 확인되었다(그림 3).
Average of culturable aerobic and anaerbic bacteria concentration in surface water, hyporheic zone water and ground water.
3.2 지표수-지하수 혼합구간의 호기 및 혐기성 세균의 분리 동정
총 24개의 지표수, 혼합구간 및 지하수 시료로 부터 호기성 세균 152개, 혐기성 세균 85개를 분리하였다. 이 중 혼합구간에서 호기성 세균 131개 집락, 혐기성 세균 70개 집락으로 총 201개의 집락을 분리하였다. 지표수는 호기성 9개 및 혐기성 7개 집락, 지하수는 호기성 12개 및 혐기성 8개 집락을 분리하였다(표 2). 특히 2016년 8월 5일에 채취한 시료에서 분리된 호기성 및 혐기성세균의 수는 2016년 12월 13일에 채취한 시료에서 분리한 세균 수 보다 상대적으로 많았다. 시료 채취 지점의 배양 가능한 미생물은 연구지 시료의 출처뿐만 아니라 계절에도 많은 영향을 받고 있었다. 분리된 집락의 동정을 위해 27F, 1492R 프라이머를 이용하여 16S rRNA 유전자를 PCR 하였고, 염기서열 분석을 진행한 결과 혼합구간에서 호기성 세균 40개 속(표 3), 혐기성 세균 21개 속(표 4)이 동정되었으며, 지표수의 경우 호기성 세균 13개 속, 혐기성 세균 5개 속이 동정 되었다. 한편 지하수의 경우 호기성 세균 5개 속, 혐기성 세균 3개 속이 동정되었다. 이 중 혼합구간에서만 나타난 속은 호기성 세균 31개 속 및 혐기성 16개 속으로 확인되었다. 혼합구간의 호기성 세균은 Pseudomonas속(약 29.3%) 및 Flavobacteriaum속(약 15.3%) 세균이 우점 하는 것으로 확인되었으며, 종 수준에서 확인한 결과 혼합구간의 우점종은 Pseudomonas extremoreintalis 및 Pseudomonas fragi가 약 5.3%로 우점 하는 것으로 확인되었다. Pseudomonas속은 다양한 환경에 존재하는데 특히 농업 토양, 그리고 식물 근권에서 자주 동정된다. 이 속은 오염물, 이물질을 분해하는 능력이 있다(Mazzola et al., 1992). 토양 건강 유지에 중요한 역할을 하는 많은 대사산물을 생산하여 병원균들로부터 주변 생태계를 보호하는 역할을 하여 환경적으로 중요하다(Tambong et al., 2017). Egamberdieva et al. (2013)의 연구결과에 따르면 Pseudomonas extremoreintalis는 염분토양과 같은 극한 환경에서도 생존할 수 있으며 주변 식물의 성장에 긍정적인 영향을 미친다고 하였다. 연구지역의 지하수는 과도한 퇴비 및 화학비료사용으로 오염이 진행되고 있으며, 겨울철 제설제 사용이 빈번하다(Kim et al., 2015). 혼합구간에서 우점하는 Pseudomonas extremoreintalis는 연구지 수환경 생태계에 긍정적인 영향을 미칠 수 있는 세균으로 판단된다.

Identification of isolation bacteria at aerobic condition from surface water, hyporheic water and groundwater.

Identification of isolation bacteria at anaerobic condition from surface water, hyporheic water and groundwater.
Pseudomonas fragi는 석유탄화수소 오염물질의 생물학적 분해가 가능한 세균이며(Adelowo et al., 2006) 황산염 환원 박테리아이기도 하다(Jayaraman et al., 1999). 황산염 환원미생물은 폐수의 황산염 뿐 만 아니라 고농도의 질산성 질소 화합물의 탈질 작용에도 영향을 미친다. Kim et al. (2018a)에 의하면 미생물과 동위원소자료 통합 분석을 통해 연구지의 혼합구간에서 탈질작용이 일어나고 있음을 확인하였다. Pseudomonas fragi는 혼합구간의 탈질작용에 영향을 미치는 세균으로 판단할 수 있다. 혼합구간의 혐기성 세균은 Pseudomonas속(약 51.4%) 및 Rahnella속(15.3%)세균이 우점 하는 것으로 확인되었으며, 종 수준에서 확인한 결과 혼합구간의 우점종은 Pseudomonas fragi가 약 17.1%로 우점 하는 것으로 확인되었다(표 3, 4).
4. 결 론
본 연구에서는 지표수, 혼합구간 및 지하수에 서식하는 배양성 미생물을 확인하였다. 이를 위해 채취한 시료로부터 미생물을 분리 및 16S rRNA 유전자 PCR을 통해 동정하였다. 배양성 미생물의 세균수를 측정한 결과, 혼합구간에서 2.5×105 CFU/ml로 지표수와 지하수에 비해 10배 이상의 세균수가 확인되었다. 호기성, 혐기성 미생물을 분리하여 세균수를 측정한 결과, 혐기성 세균보다 호기성 세균이 10-100배 더 많이 확인되었으며, 혼합구간의 호기성 미생물이 2.0×105 CFU/ml로 가장 많았다. 반면 지하수의 경우, 혐기성 미생물이 3.2×102 CFU/ml로 가장 적게 확인되었다. 혼합구간별 호기성 및 혐기성 세균수를 측정한 결과, 호기성 세균은 2.2×104– 5.6×105 CFU/ml, 혐기성 세균은 1.3×103– 6.5×104CFU/ml로 확인되었고, P6 시료에서 가장 많은 세균수가 확인되었다. 반면 혐기성 미생물은 상대적으로 P1, P3, P5 및 P6 시료에서 높게 나타난 것을 확인하였다.
호기성 및 혐기성 세균은 혼합구간의 호기성 세균 131개 집락, 혐기성 세균 70개 집락을 포함하여 총 237개 집락을 분리하였으며, 16S rRNA 유전자 염기서열 분석을 통한 동정 결과 혼합구간 호기성 세균 40개 속, 혐기성 세균 21개 속으로 동정 되었으며, 호기성 세균은 속 수준에서 Pseudomonas속이 약 29.3%로 우점하였고, Flavobacterium속이 약 15.3%로 차우점 하였다. 종 수준에서는 Pseudomonas extremorientalis 및 Pseudomonas fragi가 약 5.3%로 우점하였다. 한편 혐기성 세균은 속 수준에서 Pseudomonas속이 약 51.4%로 우점하였고, Rahnella속이 약 7.1%로 차우점 하였다. 종 수준에서는 Pseudomonas fragi가 약 17.1%로 우점하였다. 지표수, 지하수에서 분리된 속을 제외하고, 혼합구간에서만 나타난 속은 총 호기성 31개, 혐기성 16개 속으로 확인되었으며, 종 수준에서는 호기성 P. extremorientalis 포함 77종, 혐기성 P. fragi 포함 33종으로 확인되었다.
본 연구에서는 최근 많은 연구자들이 이용하는 차세대염기서열 분석 방법 중 하나인 파이로시퀀싱 방법의 한계점을 극복하고자 배양성 실험을 진행하였다. 파이로시퀀싱 방법으로 획득한 메타지노믹 자료와 본 실험을 통해 획득한 배양성 미생물과 비교분석 하면 지표수-지하수 혼합구간에 서식하는 미생물들의 생리적인 특성 및 기능과 역할에 대해 보다 명확한 기초 자료를 제공할 수 있을 것으로 여겨진다.
Acknowledgments
이 성과는 산업통상자원부의 재원으로 에너지국제공동연구사업(한국에너지기술평가원)의 지원을 받아 수행된 연구입니다(No. 20168510050070).
References
- Adelowo, O.O., Alagbe, S.O., and Ayandele, A.A., (2006), Time-dependent stability of used engine oil degradation by cultures of Pseudomonas fragi and Achromobacter aerogenes, African Journal of Biotechnology, 5(24), p2476-2479.
-
Bae, J.W., (2011), Recent methodological approaches to human microbiome, Journal of Bacteriology and Virology, 41(1), p1-7.
[https://doi.org/10.4167/jbv.2011.41.1.1]

-
Barlocher, F., and Murdoch, J.H., (1989), Hyporheic biofilms: a potential food source for interstitial animals, Hydrobiologia, 184, p61-67.
[https://doi.org/10.1007/bf00014302]

-
Brunke, M., and Gonser, T., (1997), The ecological significance of exchange processes between rivers and ground water, Freshwater Biology, 37, p1-33.
[https://doi.org/10.1046/j.1365-2427.1997.00143.x]

- Egamberdieva, D., Jabborova, D., and Mamadalieva, N., (2013), Salt tolerant Pseudomonas extremorientalis able to stimulate growth of Silybum marianum under salt stress, Medicinal and Aromatic Plant Science and Biotechnology, 7(7), p10.
- Feris, K., Ramsey, P., Frazar, C., Moore, J.N., Gannon, J.E., and Holben, W.E., (2003a), Differences in hyporheic-zone microbial community structure along a heavy-metal contamination gradient, Applied and Environmental Microbiology, 69, p5563-5573.
- Feris, K.P., Ramsey, P.W., Frazar, C., Rilling, M.C., Gannon, J.E., and Holben, W.E., (2003b), Structure and seasonal dynamics of hyporheic zone microbial communities in free-stone rivers of the western United States, Microbial Ecology, 46, p200-215.
- Graham, E.B., Crump, A.R., Resch, C.T., Fansler, S., Arntzen, E., Kennedy, D.W., Fredrickson, J.K., and Stegen, J.C., (2016), Deterministic assembly processes govern seasonal and spatial variation in microbiomes across 4 hydrologically-connected hyporheic zones, Environmental Microbiology, 19, p1552-1567.
- Grimm, N.B., and Fisher, S.G., (1984), Exchange between surface and interstitial water: implications for stream metabolism and nutrient cycling, hydrobiologia, 111, p219-228.
-
Hendricks, S.P., (1993), Microbial ecology of the hyporheic zone: a perspective intergrating hydrology and biology, Journal of the North American Benthological Society, 12, p70-78.
[https://doi.org/10.2307/1467687]

-
Huang, Y.F., Chen, S.C., Chiang, Y.S., Chen, T.H., and Chiu, K.P., (2012), Palindromic sequence impedes sequencing-by-ligation mechanism, BMC Systems Biology, 6(2), pS10.
[https://doi.org/10.1186/1752-0509-6-s2-s10]

-
Iwamoto, T., Tani, K., Nakamura, K., Suzuki, Y., Kitagawa, M., Eguchi, M., and Nasu, M., (2000), Monitoring impact of in situ biostimulation treatment on groundwater bacterial community by DGGE, FEMS Microbiology Ecology, 32(2), p129-141.
[https://doi.org/10.1016/s0168-6496(00)00022-2]

-
Jayaraman, A., Mansfeld, F.B., and Wood, T.K., (1999), Inhibiting sulfate-reducing bacteria in biofilms by expressing the antimicrobial peptides indolicidin and bactenecin, Journal of Industrial Microbiology and Biotechnology, 22(3), p167-175.
[https://doi.org/10.1038/sj.jim.2900627]

-
Jeon, W.H., Park, S., Lee, J.Y., Lee, J.S., and Cho, Y.J., (2018), Evaluation of the groundwater and stream water interaction using metagenomics analysis in the hyporheic zone, Inje of Gangwon Province, Journal of the Geological Society of Korea, 54(3), p293-300, (in Korean with English abstract).
[https://doi.org/10.14770/jgsk.2018.54.3.293]

- Jones, J.B., Fisher, S.G., and Grimm, N.B., (1995), Nitrification in the hyporheic zone of a desert stream ecosystem, Journal of the North American Benthological Society, 14, p249-258.
- Kim, J.J, Jeon, W.H., and Lee, J.Y., (2018b), Estimation of groundwater recharge using field moisture observations and HYDRUS-1D modeling in Haean basin, Journal of the Geological Society of Korea, 54(5), p545-556, (in Korean with English abstract).
-
Kim, H., Kaown, D., Mayer, B., Lee, J.Y., Hyun, Y., and Lee, K.K., (2015), Identifying the sources of nitrate contamination of groundwater in an agricultural area (Haean basin, Korea) using isotope and microbial community analyses, Science of the Total environment, 533, p566-575.
[https://doi.org/10.1016/j.scitotenv.2015.06.080]

- Kim, H., Kaown, D., Mayer, B., Lee, J.Y., and Lee, K.K., (2018a), Combining pyrosequencing and isotopic approaches to assess denitrification in a hyporheic zone, Science of the Total Environment, 631, p755-764.
- Kim, H., and Lee, K.K., (2019), Effect of vertical flow exchange on microbial community distributions in hyporheic zones, Episodes, 42(1), p1-16.
-
Kim, H., Lee, K.K., and Lee, J.Y., (2014), Numerical verification of hyporheic zone depth estimation using streambed temperature, Journal of Hydrology, 511, p861-869.
[https://doi.org/10.1016/j.jhydrol.2014.02.052]

-
Lee, J.H., Lee, B.J., and Unno, T., (2018), Bacterial communities in ground-and surface water mixing zone induced by seasonal heavy extraction of groundwater, Geomicrobiology Journal, 35, p768-774.
[https://doi.org/10.1080/01490451.2018.1468834]

- Lee, J.Y., (2009), Importance of hydrogeological and hydrologic studies for Haean basin in Yanggu, Journal of the Geological Society of Korea, 45(4), p405-414, (in Korean with English abstract).
-
Lee, J.Y., Raza, M., and Kwon, K.D., (2019), Land use and land cover changes in the Haean Basin of Korea: Impacts on soil erosion, Episodes, 42(1), p17-32.
[https://doi.org/10.18814/epiiugs/2019/0190003]

- Mazzola, M., Cook, R.J., Thomashow, L.S., Weller, D.M., and Pierson, L.S., (1992), Contribution of phenazine antibiotic biosynthesis to the ecological competence of fluorescent pseudomonads in soil habitats, Applied and Environmental Microbiology, 58(8), p2616-2624.
- Mugani, R., Messana, G., and Di Lorenzo, T., (2015), The hyporheic zone and its functions: revision and research status in Neotropical regions, Brazilian Journal of Biology, 75, p524-534.
-
Murray, I.A., Clark, T.A., Morgan, R.D., Boitano, M., Anton, B.P., Luong, K., Fomenkov, A., Turner, S.W., Korlach, J., and Roberts, R.J., (2012), The methylomes of six bacteria', Nucleic Acids Research, 40(22), p11450-11462.
[https://doi.org/10.1093/nar/gks891]

-
Quail, M.A., Smith, M., Coupland, P., Otto, T.D., Harris, S.R., Connor, T.R., and Gu, Y., (2012), A tale of three next generation sequencing platforms: comparison of Ion Torrent, Pacific Biosciences and Illumina MiSeq sequencers, BMC genomics, 13(1), p341.
[https://doi.org/10.1186/1471-2164-13-341]

-
Storey, R.G., Fulthorpe, R.R., and Williams, D.D., (1999), Perspectives and predictions on the microbial ecology of the hyporheic zone, Freshwater Biology, 41, p119-130.
[https://doi.org/10.1046/j.1365-2427.1999.00377.x]

- Tambong, J.T., Xu, R., and Bromfield, E.S., (2017), Pseudomonas canadensis sp. nov., a biological control agent isolated from a field plot under long-term mineral fertilization, International Journal of Systematic and Evolutionary Microbiology, 67(4), p889-895.
-
Triska, F.J., Duff, J.H., and Avanzino, R.J., (1993), The role of water exchanage between a stream channel and its hyporheic zone in nitrogen cycling at the terrestrial aquatic interface, Hydrobiologia, 251, p167-184.
[https://doi.org/10.1007/bf00007177]

-
Pei, Y.S., Wang, J., Wang, Z.Y., and Yang, Z.F., (2010), Characteristics of Ammonia-oxidizing and Denitrifying Bacteria at the River-sediment Interface, Procedia Environmental Sciences, 2, p1988-1996.
[https://doi.org/10.1016/j.proenv.2010.10.214]

- Pusch, M., and Schwoerbel, J., (1994), Community respiration in hyporheic sediments of a mountain stream (Steina, Black Forest), Archiv für Hydrobiologie, 130, p35-52.
-
van Vliet, A.H., (2010), Next generation sequencing of microbial transcriptomes: challenges and opportunities, FEMS Microbiology Letters, 302(1), p1-7.
[https://doi.org/10.1111/j.1574-6968.2009.01767.x]

- Won, J.K., Lee, M.W., and Woo, J.K., (1987), Geologic and geographic characteristics of Haean basin, Memorial Book of 40th Anniversary of Kangwon Nation University Kangwon National University Publishing, Chuncheon, p45-57.
- Yoon, S.H., Ha, S.M., Kwon, S., Lim, J., Kim, Y., Seo, H., and Chun, J., (2017), Introducing EzBioCloud: A taxonomically united database of 16S rRNA and whole genome assemblies, International Journal Of Systematic And Evolutionary Microbiology, 67, p1613-1617.