강원도 인제군 혼합대에서 메타지놈 분석을 통한 지하수-하천수 상호작용 평가
초록
본 연구는 강원도 인제군 인북천 혼합대에서 생물학적인 방법을 이용하여 지하수-하천수 상호작용을 평가하였다. 메타지놈 분석은 원핵 미생물과 진핵 미생물 분석으로 구성된다. 분석을 위해 2016년 12월 2일 인북천에 10 m 간격으로 설치된 4개의 피에조미터(IYHW1, IYHW2, IYHW3, IYHW4)에서 혼합대 물시료를 채취하였으며 대조군으로 하천수(IYSW1)와 인근 농업용 관정(IYAW1)에서 지하수 시료를 채취하였다. 또한 수리화학적 특성을 파악하기 위해 현장수질 분석과 양·음이온 분석을 수행하였다. 원핵 미생물의 군집 구조는 Proteobacteria가 하천수인 IYSW1과 IYHW1을 제외한 모든 시료에서 90.5~96.8%로 높은 비율을 보였다. 또한 진핵 미생물의 군집 구조는 Chytridiomycota 문은 IYSW1과 IYHW1에서 각각 57.8과 43.4%의 높은 비율을 차지하는 반면 IYAW1과 IYHW4에서는 0.2% 이하로 거의 존재하지 않았다. 전반적으로 IYSW1과 IYHW1은 하천의 영향을 우세하게 받았고 IYAW1과 IYHW4는 지하수의 영향을 우세하게 받아 서로 유사한 미생물 군집 구조를 보였다. 그리고 IYHW2와 IYHW3은 지하수-하천수 상호작용의 점이지대로 판단된다.
Abstract
This study was evaluated for groundwater and stream water interaction in the hyporheic zone of Inbuk Stream, Inje of Gangwon Province, using biological methods. A metagenomic analysis consists of eukaryotic microbial and fungi microbial communities. For this purpose, the samples of hyporheic water were collected from the four piezometers (IYHW1, IYHW2, IYHW3, and IYHW4) that were installed at 10 m interval across the stream. As a control group, the stream water (IYSW1) and the groundwater (IYAW1) were sampled on December 2, 2016. We also measured field parameters and analyzed chemical compositions of the hyporheic waters, stream water, and groundwater. The structure of prokaryote microbial community for high rates of Proteobacteria showed a high rate (90.5~96.8%) for all samples except IYSW1 and IYHW1. Also, Chytridiomycota in the structure of eukaryotic microbial community was predominant in IYSW1 (57.8%) and IYHW1 (43.4%) but was rarely (<0.2%) found in IYAW1 and IYHW4. As a result, it is inferred that IYSW1 and IYHW1 were dominated by stream water, and IYAW1 and IYHW4 were found to be dominated by groundwater. And IYHW2 and IYHW3 were a point zone of interaction between groundwater and stream water, where microbial and hydrological changes were dynamic.
Keywords:
metagenomics, prokaryote, eukaryote, biological indicator, hyporheic zone, groundwater and stream water interaction키워드:
메타지놈, 원핵 미생물, 진핵 미생물, 생물학적 지시, 혼합대, 지하수-하천수 상호작용1. 서 론
최근 많은 나라에서 온도 상승, 해수면 상승, 생태계 변화, 이상기후 등에 따른 물수지 변화로 지하수 및 하천수 자원의 안정적인 확보가 어려워지고 있으며 이에 따라 수자원의 중요성이 점차 부각되면서 자원으로서 물을 관리하고 지속적으로 이용하기 위한 노력들이 많은 연구자들에 의해 이루어지고 있다(Jyrkama and Sykes, 2007; Herrera-Pantoja and Hiscock, 2008; Allen et al., 2010). 또한 기후변화에 따른 지하수와 하천수의 수량 및 수질 변화는 직·간접적으로 접경구역의 변화를 가져오며 주변 생태계에도 영향을 준다.
지하수와 하천수의 접경구역인 혼합대(hyporheic zone)는 두 수체간 물과 물질 교환이 역동적으로 발생하는 경계면(interface)이다(Brunke and Gonser, 1997; Winter et al., 1998; Kalbus et al., 2006). 지하수와 하천수는 서로 밀접하게 연결되어 있으며 상호 영향을 주는 것으로 파악되어 수리적, 화학적, 열적, 생물학적 등 다양한 접근 방법을 통해 연구되어지고 있다(Winter et al., 1998; Anderson, 2005; Bourke et al., 2014; Kim et al., 2014; Yang et al., 2014; Jeon et al., 2015). 일반적으로 우기에는 지하수위가 하천수위보다 낮아 하천수가 유입되어 지하수를 충진시키는 손실하천을 보이는 반면 건기에는 지하수위가 하천수위보다 높아 포화대로부터 지하수가 배출되어 하천유량을 유지시키는 이득하천을 보인다(Sophocleous, 2002).
최근에는 생물학적인 방법을 이용하여 지하수-하천수의 상호작용을 평가하고 있으며 특히 하천 내 서식하고 있는 미생물에 대한 연구가 진행되고 있다(Kim et al., 2018). 하지만 대부분 세균(Bacteria)과 고세균(Archaea)으로 이루어진 원핵 미생물에 초점이 맞추어 있다. 진핵 미생물도 생태계 전반에 서식하고 있으며 다양한 생화학적 순환에 있어서 중요한 역할을 하고 있기 때문에 진핵 미생물에 대해 초점을 두고 연구를 수행할 필요가 있다(Hindshaw et al., 2017). 이처럼 다양한 생리적 기능을 가지고 있는 미생물에 대하여 연구하는 것은 하천의 오염물질 분해와 탄소, 질소, 인, 황과 같은 주요 원소뿐만 아니라 미량 원소의 순환에 있어서 중요하다. 이에 따라 지하수, 하천수, 혼합대에서 어떤 종류의 미생물이 주로 서식하고 있으며 특정 미생물의 어떤 유전자가 하천 생태계에 중요한 영향을 미치는지 분석할 필요가 있다(Baker et al., 2015; Dopheide et al., 2015). 본 연구에서는 강원도 인제군 서화면 인북천에서 지하수, 하천수, 혼합대수의 메타지놈 분석을 통해 원핵 미생물 및 진핵 미생물의 군집 구조를 파악하고 지하수-하천수 상호작용을 평가하였다.
2. 연구 방법
2.1 연구지역 및 시료채취
연구지역은 소양강 상류유역으로 행정구역상 강원도 인제군 서화면 양지교 아래 흐르는 인북천이며 지리적 방위는 동경 128o12’24”, 북위 38o13’16”이다(그림 1). 연구지역의 북쪽에는 양구 해안분지(펀치볼)와 접하고 있으며 대체로 동부와 서부의 지세가 높고 남북방향으로 하천이 발달되어 있는 지형을 보인다. 연구지역 일대의 일반지질은 고원생대 변성암인 흑운모편마암, 함석류석 화강편마암, 대암산 석영섬장암 등이 주를 이루고 이를 중생대 쥬라기의 섬록암, 흑운모화강암, 반상 흑운모화강암이 관입한다. 또한 이들을 제4기 충적층이 부정합으로 덮는다(Song and Cho, 2009).
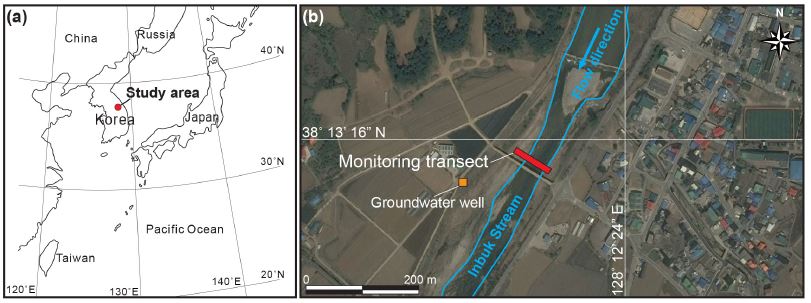
Map of the study area (Inbuk stream) showing the groundwater well and sampling transect for stream water and hyporheic water.
하천 좌측인 북서 방향에 약 10 m 간격으로 4개의 피에조미터(IYHW1, IYHW2, IYHW3, IYHW4)가 지하수-하천수 접경구역인 혼합대에 설치되었으며 스크린 길이는 약 8 cm이다(그림 2). 연구지역에서 하류방향으로 90 m 떨어진 지점에 설치심도 72 m의 대형 농업용 관정(IYAW1)이 위치한다. 물 시료는 2016년 12월 2일에 농업용 관정, 하천수(IYSW1), 4지점의 피에조미터에서 연동펌프(Peristaltic pump; Model 410, Solinst)를 이용하여 채취하였으며 농업용 관정과 피에조미터 내에 고여 있는 물은 주변의 수질특성과 다르기 때문에 적정량(일반적으로 관정 내 지하수량의 3~5배) 양수한 후 채취하였다.
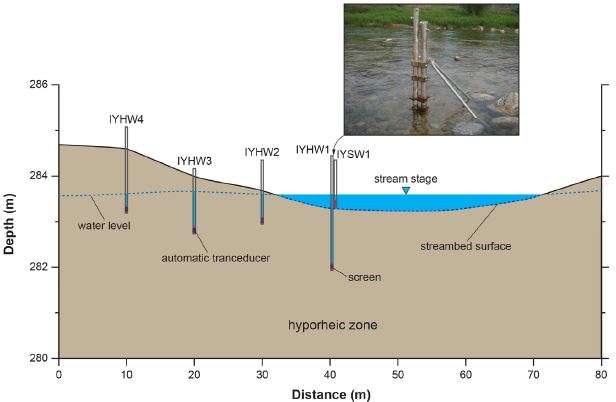
Vertical cross sections of four piezometers (IYHW1, IYHW2, IYHW3, IYHW4) showing stream stage, water level, streambed surface and automatic data loggers (water level and temperature).
미생물 분석을 위해 각 지점마다 35 L씩 무균채수병에 채취하였다. 또한 현장수질측정과 함께 양·음이온 분석을 위해 채취된 시료를 0.45 µm 필터로 여과하였으며 분석항목에 따라 전처리과정을 거친 후 시료채취 용기에 담아 강원도 원주시에 위치한 상지대학교 자연과학연구지원센터에 분석 의뢰하였다. 시료 채취 시 사용된 호스는 교차오염을 방지하기 위해 증류수를 사용해 세척하였으며 시료 운반 시 아이스박스에 4℃ 내외로 보관하였다. 이날 강수는 0 mm, 최고기온 7.9, 최저기온 –3.5, 평균기온은 0℃를 기록하였으며 강수량 10 mm 이하 무강수 일수는 11월 1일부터 12월 1일까지 31일 이었다(KMA, 2016).
2.2 DNA 추출
농업용 관정에서 채취한 지하수와 하천수 시료를 혼합대에 대한 대조군으로 사용하였으며 혼합대수를 포함하여 총 6개 시료에 대해 메타지놈 분석을 수행하였다. 지하수, 하천수 및 혼합대서 각각 채취한 35 L의 물 시료를 0.25 µm 이하로 여과하였다. 필터 위에 걸러진 물질들을 긁개를 이용해 1.5 ml 튜브에 옮긴 후 DNA 추출(FastDNATM SPIN Kit, MP Biomedicals)을 수행하였다. 이를 위해 시료를 비드(bead)가 들어 있는 튜브로 옮기고 용균 완충액(Lysis buffer solution)을 넣은 후 4℃에서 30분 동안 균질화 하였다. 이후 원심분리기를 12,000 rpm으로 10분 동안 작동하여 상등액에서 단백질을 침전시켰고 DNA binding silica resin과 결합시켰으며 필터링 스핀 튜브(filtering SPIN tube)로 옮겨주는 과정을 반복하였다. 마지막 단계에서 DNA를 DES (DNase/Pyrogen-free water) 버퍼로 새 튜브에 용출시켰다.
2.3 PCR 증폭과 Illumina sequencing 방법
PCR 증폭을 위해 원핵 미생물은 16S rRNA 유전자의 V3-V4 region 부분이 태깅된 341F와 805R 프라이머를 이용하였고 진핵 미생물은 ITS2 부분의 fITS7과 ITS4 프라이머를 이용하였다(Ihrmark et al., 2012). PCR 증폭 과정에서 NEB Phusion Hot Start Flex 2X Master Mix (M0536S, NEB, UK)를 사용하였다. 초기 변성(denaturation) 증폭반응 조건은 1단계 98℃에서 30초 수행하였고 2단계 98℃에서 10초, 55℃에서 30초, 72℃에서 30초 과정으로 chimeric 염기서열 형성을 최대한 억제하기 위해 25회 수행하였으며 마지막 3단계 72℃에서 5분 과정을 수행하였다. 특히 annealing 과정에서 ramp speed를 1℃/초로 제한하였다. 증폭된 결과물은 전기영동으로 확인 및 정량화한 후 동일 농도로 pooling하였다. 준비된 amplicon 100 ng으로부터 NEB Ultra II DNA Library Prep Kit for Illumina (E7645S, NEB, UK)를 이용하여 sequencing library를 제작하였으며 제작 과정은 제조사의 프로토콜에 따라 천랩(ChunLab Inc.)에서 수행되었다.
2.4 MiSeq pipeline system
MiSeq sequencing 결과물에 대한 품질검사를 위해 Trimmomatic 프로그램(Bolger et al., 2014)을 사용하였으며 품질검사를 통과한 서열 데이터는 다시 Pandseq 프로그램(Masella et al., 2012)을 사용하여 Paired-End 서열에 대한 분석을 수행하였다. 또한 Miseq sequencing 분석 과정 중에 발생하는 기계적 또는 분석원리상의 무작위적 오류를 제거하기 위해 DUDE-Seq 프로그램(Lee et al., 2017)을 사용하였다. 여러 단계를 통해 정제된 높은 품질의 서열 데이터를 이용하여 분류학적인 동정(Taxonomic assignment)을 수행하였다. 군집분석을 위해 EzBioCloud의 동정 알고리즘을 응용하였다(www.ezbiocloud.net). PCR 증폭 과정에서 생긴 키메라(chimera)서열을 탐지하기 위해 Uchime 6 (Edgar et al., 2011)와 EzTaxon에서 제공되는 non-chimeric 데이터베이스를 이용하였다. 이때 약 97% 이상의 유사도를 보이는 서열은 동정단계에서 이미 종 수준으로 분류되었으므로 탐지대상에서 제외시켰다. 이렇게 분석된 서열 데이터는 CD-Hit 7 (Fu et al., 2012)과 UCLUST 8 프로그램(Edgar, 2010)을 이용하였으며 원핵 미생물에 대한 군집분석은 천랩(ChunLab Inc.)에서 수행하였고 진핵 미생물에 대한 군집분석은 극지연구소에서 수행하였다.
3. 결과 및 토의
3.1 일반 수리화학적 특성
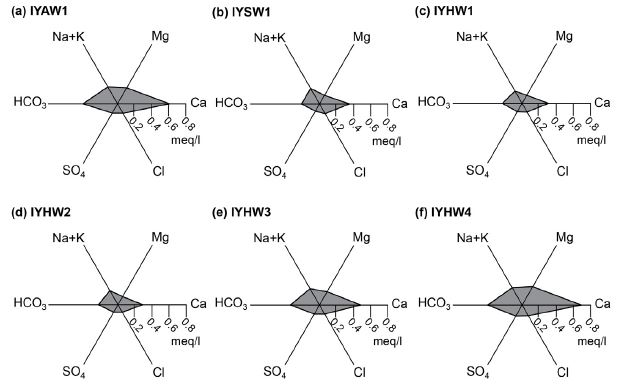
표 1은 연구지역의 지하수, 하천수, 혼합대수의 수리화학적 특성에 대한 결과를 보여준다. pH는 6.5~7.8의 범위를 보였으며 농업용 관정인 IYAW1이 6.5로 가장 낮았고 IYHW2이 7.8로 가장 높았다. 전기전도도는 하천으로부터 비교적 멀리 위치한 IYHW3, IYHW4, IYAW1로 갈수록 높게 나타나 하천수보다 지하수의 영향을 더 많이 받은 것으로 판단된다. 용존산소(DO)는 3.63~7.42 mg/l의 범위를 보였으며 지하수가 3.63 mg/l로 가장 낮았고 하천수가 7.42 mg/l로 가장 높았다. 그림 3에서는 양·음이온에 대한 수질 유형을 스팁다이어그램을 통해 나타내었다. 본 연구지역의 양이온은 Ca>Na>Mg>K 순으로 나타났고 음이온은 HCO3>NO3>SO4>Cl 순으로 나타났다. 용존성분들 중 Ca와 HCO3가 가장 우세하게 나타나 일반적인 천부 지하수 및 하천수 환경을 지시한다(Hem, 1985). 또한 하천과 하천에서 가까운 피에조미터에서는 낮은 Ca와 HCO3 농도를 보이지만 전기전도도와 마찬가지로 하천으로부터 멀리 위치한 IYHW3, IYHW4, IYAW1로 갈수록 농도가 점차 높아지는 것을 볼 수 있다.

Field measured parameters and ionic compositions of groundwater, hyporheic water and stream water on December 2, 2016.
3.2 원핵 미생물과 진핵 미생물의 군집분석
그림 4는 원핵 미생물 군집 분석결과이며 문(phylum) 수준에서 보이는 경향성을 종(species) 수준에서 비교하기 위한 분류구성(taxonomic composition)이다. 더블파이 차트의 내부 파이는 문 수준이며 외부 파이는 Proteobacteria만을 대상으로 하는 종 수준을 나타내어 시료(위치) 간의 원핵 미생물 군집구조의 관계를 확인하였다. 또한 외부 파이의 종은 수 천 가지 이상이기 때문에 가장 높은 비율을 보이는 종 15개 종을 선별하였으며 이외의 종은 기타(ETC)로 처리하였다. 모든 시료에서 가장 높은 비율을 차지하는 Proteobacteria는 일반적으로 물에 많이 존재하는 것으로 알려진 박테리아 문이다. 하천수인 IYSW1과 IYHW1을 제외한 나머지 지하수 및 혼합대의 시료에서 90.5~96.8%의 Proteobacteria가 포함되어 있다. 하천수인 IYSW1에서는 53.7%의 Proteobacteria가 포함되어 있었으며 IYSW1과 동일한 위치인 IYHW1의 Proteobacteria는 82.9%로 지하수보다 하천수의 영향을 더 받은 것으로 판단된다. 지하수와 혼합대에서는 Proteobacteria가 대부분 차지하고 있지만 하천수에서는 Proteobacteria 이외에도 Bacteroidetes (22.4%)와 Verrucomicrobia (16.1%)가 함께 포함되어 있다. 특히 하천의 중심부와 가까운 IYHW1과 IYHW2에서 Bacteroidetes의 포함 비율은 각각 1.7%와 1.4%로 지하수 또는 나머지 혼합대에서의 시료(1% 미만)보다 다소 높았다.
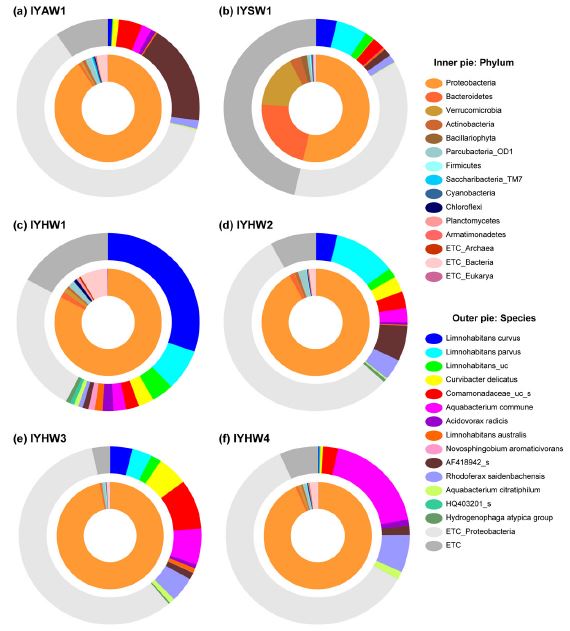
Double pie chart showing the taxonomic composition at phylum and species level for groundwater (IYAW1), hyporheic water (IYHW1, IYHW2, IYHW3, and IYHW4) and stream water (IYSW1) on December 2, 2016.
외부 파이차트인 종 수준의 Limnohabitans curvus, Limnohabitans parvus, Limnohabitans_unclassified는 IYSW1, IYHW1, IYHW2, IYHW3에서 높은 구성 비율(각각 41.7, 16.6, 9.3, 10.8%)을 보인 반면 IYHW4와 IYAW1에서는 1% 미만의 값을 보였다. 특히 Limnohabitans는 지하수보다 하천수의 영향을 더 많이 받는 미생물 종으로 판단된다. 또한 Aquabacterium commune와 AF418942_s는 IYHW4와 IYAW1에서 다른 지점의 시료들 보다 높은 비율을 보여 하천수보다 지하수의 영향을 더 많이 받는 종으로 판단되며 실제로 IYHW4 피에조미터 방향으로 갈수록 하천과 멀리 떨어진다. 위 5개의 미생물 종이 가지고 있는 특별한 효소 또는 단백질이 혼합대에서 어떠한 역할을 할 수 있는지 확인하기 위해 이들이 가지고 있는 유전자 정보를 확인하였으나 Limnohabitans curvus와 Limnohabitans parvus를 제외한 나머지 종들에서는 이들의 완벽한 유전자 정보를 확인할 수 없었다. 이를 확인하기 위해서는 실제로 미생물을 채집하여 whole-genome sequencing을 수행해야 한다.
진핵 미생물 군집분석 결과 Chytridiomycota 문이 IYSW1과 IYHW1에서 각각 57.8과 43.4%의 비율로 높게 나타났고 특히 Rhizophydiales 목(order)이 속하였다. 하지만 IYAW1과 IYHW4에는 0.2% 이하로 거의 존재하지 않아 Chytridiomycota 문은 지하수보다 하천수의 영향을 더 많이 받는 미생물 군임을 판단 할 수 있다(그림 5). Ascomycota 문은 IYAW1과 IYHW4에서는 각각 10.0과 10.5%의 비율을 보였고 하천과 하천에서 비교적 가까운 피에조미터에서는 14.6~43.9% 범위를 보였다. 또한 진핵 미생물 군집분석 결과에서 상당 부분을 차지하고 있는 Fungi_unclassified의 경우 계(Kingdom)는 Fungi에 속하나 그 하위 단계에서부터는 분류할 수 없는 Fungi에 속해 있는 것을 확인할 수 있다.
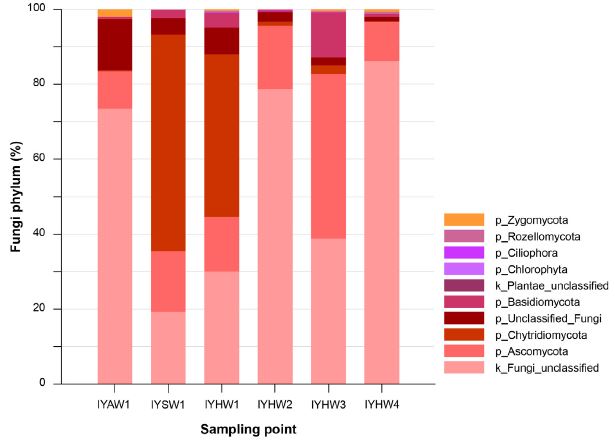
Fungi phylum for groundwater (IYAW1), hyporheic water (IYHW1, IYHW2, IYHW3, and IYHW4) and stream water (IYSW1) on December 2, 2016 (p: phylum, k: kingdom).
추가적으로 혼합대에서만 특이적으로 살고 있는 진핵 미생물 중 Lobulomyces는 특히 IYHW3에서 발견되는데 이 종은 대부분의 Fungi류가 가지고 있는 키틴질(chitin)의 세포벽이 없는 섭식영양 기생충(phagotrophic parasite)으로 다른 세포 내로 들어갈 수 있는 특징을 가지고 있다(Held, 1981; Powel, 1984; Stajich et al., 2009). 세포벽에 키틴질이 없는 호상균(chytrid) 중 하나이며 소엽 모양(lobular shape)을 가지고 있어 현재까지 밝혀진 Lobulomyces의 종은 단 두 종뿐이다(Simmons et al., 2009). 이렇게 유일하게 혼합대에만 존재하는 진핵 미생물은 혼합대에서의 하천수 또는 지하수의 영향을 평가할 수 있는 생물학적 지표로 사용될 수 있음을 제시한다.
4. 결 론
본 연구는 인북천 혼합대에서 지하수, 하천수, 혼합대수의 메타지놈 분석을 통해 미생물 군집 구조를 확인하였다. 전반적으로 IYSW1과 IYHW1은 하천수 환경에 가깝고 IYAW1과 IYHW4는 지하수 환경에 가까워 일반 수리화학적 특성이나 원핵 미생물과 진핵 미생물 유형이 비슷한 것으로 나타났다. 또한 IYHW2와 IYHW3은 지하수-하천수 상호작용의 점이지대로 다이내믹한 변화를 보였다. 원핵 미생물 중 Proteobacteria가 모든 시료에서 높은 비율로 나타났지만 IYSW1과 하천에서 가까운 IYHW1에서는 각각 53.7과 82.9%로 다른 지점(90% 이상) 보다 상대적으로 낮은 비율을 보였다. 이는 Proteobacteria가 하천수보다 지하수에 영향을 받은 것으로 판단된다.
또한 혼합대에서 우점하여 존재하고 있는 5개의 미생물 종을 공통으로 가지고 있으며 혼합대에서 특이적으로 어떤 기능을 하는 효소 또는 단백질이 무엇인지 파악하기 위해 whole-genome sequencing이 필요할 것으로 판단된다. 이처럼 혼합대에서의 미생물 구성과 변화를 연구함으로써 지하수-하천수 상호작용을 정성적 또는 정량적으로 평가할 수 있는 지표로 사용될 가능성을 볼 수 있었으며 원소 순환의 모니터링을 통해 미생물과 이들이 가지고 있는 특정 유전자들이 혼합대에서 어떤 역할을 하고 있는지 규명할 수 있을 것으로 기대된다.
Acknowledgments
이 성과는 2018년도 정부(과학기술정보통신부)의 재원으로 한국연구재단의 지원을 받아 수행된 연구임(No. NRF-2015R1A4A1041105). 또한 이 논문은 2015년도 강원대학교 학술연구조성비(D1000307-01-01)의 지원을 받아 수행되었습니다.
References
-
Allen, C.D., Macalady, A.K., Chenchouni, H., Bachelet, D., McDowell, N., Vennetier, M., Kitzberger, T., Rigling, A., Breshears, D.D., Hogg, E.H., Gonzales, P., Fensham, R., Zhang, Z., Castro, J., Demidova, N., Lim, J.H., Allard, G., Running, S.W., Semerci, A., and Cobb, N., (2010), A global overview of drought and heat-induced tree mortality reveals emerging climate change risks for forests, Forest Ecology and Management, 259, p660-684.
[https://doi.org/10.1016/j.foreco.2009.09.001]

-
Anderson, M.P., (2005), Heat as a ground water tracer, Groundwater, 43(6), p951-968.
[https://doi.org/10.1111/j.1745-6584.2005.00052.x]

-
Baker, B.J., Lazar, C.S., Teske, A.P., and Dick, G.J., (2015), Genomic resolution of linkages in carbon, nitrogen, and sulfur cycling among widespread estuary sediment bacteria, Microbiome, 3, p14.
[https://doi.org/10.1186/s40168-015-0077-6]

-
Bolger, A.M., Lohse, M., and Usadel, B., (2014), Trimmomatic: a flexible trimmer for Illumina sequence data, Bioinformatics, 30(15), p2114-2120.
[https://doi.org/10.1093/bioinformatics/btu170]

-
Bourke, S.A., Cook, P.G., Shanafield, M., Dogramaci, S., and Clark, J.F., (2014), Characterization of hyporheic exchange in a losing stream using radon-222, Journal of Hydrology, 519, p94-105.
[https://doi.org/10.1016/j.jhydrol.2014.06.057]

-
Brunke, M., and Gonser, T., (1997), The ecological significance of exchange processes between rivers and groundwater, Freshwater Biology, 37, p1-33.
[https://doi.org/10.1046/j.1365-2427.1997.00143.x]

-
Dopheide, A., Lear, G., He, Z., Zhou, J., and Lewis, G.D., (2015), Functional gene composition, diversity and redundancy in microbial stream biofilm communities, PLoS ONE, 10(4), e0123179.
[https://doi.org/10.1371/journal.pone.0123179]

-
Edgar, R.C., (2010), Search and clustering orders of magnitude faster than BLAST, Bioinformatics, 26(19), p2460-2461.
[https://doi.org/10.1093/bioinformatics/btq461]

-
Edgar, R.C., Haas, B.J., Clemente, J.C., Quince, C., and Knight, R., (2011), UCHIME improves sensitivity and speed of chimera detection, Bioinformatics, 27(16), p2194-2200.
[https://doi.org/10.1093/bioinformatics/btr381]

-
Fu, L., Niu, B., Zhu, Z., Wu, S., and Li, W., (2012), CD-HIT: accelerated for clustering the next-generation sequencing data, Bioinformatics, 28(23), p3150-3152.
[https://doi.org/10.1093/bioinformatics/bts565]

-
Held, A.A., (1981), Rozella and Rozellopsis: Naked endoparasitic fungi which dress-up as their hosts, The Botanical Review, 47(4), p451-515.
[https://doi.org/10.1007/bf02860539]

- Hem, J.D., (1985), Study and interpretation of the chemical characteristics of natural water, Third Edition, United States Geological Survey, Water-Supply Paper, 2254, p263.
-
Herrera-Pantoja, M., and Hiscock, K.M., (2008), The effects of climate change on potential groundwater recharge in Great Britain, Hydrological Processes, 22, p73-86.
[https://doi.org/10.1002/hyp.6620]

-
Hindshaw, R.S., Lindsay, M.R., and Boyd, E.S., (2017), Diversity and abundance of microbial eukaryotes in stream sediments from Svalbard, Polar Biology, 40(9), p1835-1843.
[https://doi.org/10.1007/s00300-017-2106-3]

- Ihrmark, K., Bödeker, I.T.M., Cruz-Martinez, K., Friberg, H., Kubartova, A., Schenck, J., Strid, Y., Stenlid, J., Brandström-Durling, M., Clemmensen, K.E., and Lindahl, B.D., (2012), New primers to amplify the fungal ITS2 region-evaluation by 454-sequencing of artificial and natural communities, FEMS Microbiology Ecology, 82, p666-677.
-
Jeon, W.H., Lee, J.Y., Cheong, W.Y., Park, Y.H., Oh, S.H., Eum, D.H., and Park, J.Y., (2015), A multi-method approach revealing the groundwater-stream water interaction in the Inbuk stream, Korea, Geosciences Journal, 19(2), p325-340.
[https://doi.org/10.1007/s12303-014-0043-5]

-
Jyrkama, M.I., and Sykes, J.F., (2007), The impact of climate change on spatially varying groundwater recharge in the grand river watershed (Ontario), Journal of Hydrology, 338, p237-250.
[https://doi.org/10.1016/j.jhydrol.2007.02.036]

-
Kalbus, E., Reinstorf, F., and Schirmer, M., (2006), Measuring methods for groundwater-surface water interactions: a review, Hydrology and Earth System Sciences, 10, p873-887.
[https://doi.org/10.5194/hess-10-873-2006]

-
Kim, H., Kaown, D., Mayer, B., Lee, J.Y., and Lee, K.K., (2018), Combining pyrosequencing and isotopic approaches to assess denitrification in a hyporheic zone, Science of the Total Environment, p631-632, 755-764.
[https://doi.org/10.1016/j.scitotenv.2018.03.073]

-
Kim, H., Lee, K.K., and Lee, J.Y., (2014), Numerical verification of hyporheic zone depth estimation using streambed temperature, Journal of Hydrology, 511, p861-869.
[https://doi.org/10.1016/j.jhydrol.2014.02.052]

- KMA (Korean Meteorological Administration), (2016), http://kma.go.kr (June 21, 2018).
-
Lee, B., Moon, T., Yoon, S., and Weissman, T., (2017), DUDE-Seq: Fast, flexible, and robust denoising for targeted amplicon sequencing, PLoS ONE, 12(7), e0181463.
[https://doi.org/10.1371/journal.pone.0181463]

-
Masella, A.P., Bartram, A.K., Truszkowski, J.M., Brown, D.G., and Neufeld, J.D., (2012), PANDAseq: paired-end assembler for illumina sequences, BMC Bioinformatics, 13, p31.
[https://doi.org/10.1186/1471-2105-13-31]

-
Powell, M.J., (1984), Fine structure of the unwalled thallus of Rozella polyphagi in its host polyphagus euglenae, Mycologia, 76(6), p1039-1048.
[https://doi.org/10.2307/3793019]

-
Simmons, D.R., James, T.Y., Meyer, A.F., and Longcore, J.E., (2009), Lobulomycetales, a new order in the Chytridiomycota, Mycological Research, 113(4), p450-460.
[https://doi.org/10.1016/j.mycres.2008.11.019]

- Song, K.Y., and Cho, D.L., (2009), Geological report of the Mandaeri sheet scale 1:50,000, Korea Institute of Geoscience and Mineral Resources.
-
Sophocleous, M., (2002), Interactions between groundwater and surface water: the state of the science, Hydrogeology Journal, 10, p52-67.
[https://doi.org/10.1007/s10040-002-0204-x]

- Stajich, J.E., Berbee, M.L., Blackwell, M., Hibbett, D.S., James, T.Y., Spatafora, J.W., and Taylor, J.W., (2009), The Fungi, Current Biology, 19(18), pR840-R845.
- Winter, T.C., Harvey, J.W., Franke, O.L., and Alley, W.M., (1998), Ground water and surface water-a single resource, U.S. Geological Survey Circular, 1139.
-
Yang, Z., Zhou, Y., Wenninger, J., and Uhlenbrook, S., (2014), A multi-method approach to quantify groundwater/surface water-interactions in the semi-arid Hailiutu River basin, northwest China, Hydrogeology Journal, 22, p527-541.
[https://doi.org/10.1007/s10040-013-1091-z]